相関係数をRで計算する
はじめに
以前に相関係数の話をしたが、Rでの計算方法を今回あわせて書いておきたい。
mstour.hatenablog.com
Rで計算
相関係数の話で、次のような図を例に出した。
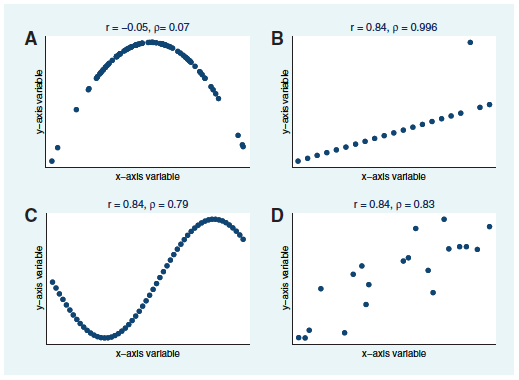
今回は同様のデータを作成して相関を計算してみる。相関の計算にはstatsパッケージの関数を使うが、これはRの起動のときに自動で読み込まれているので特別な作業はいらない。
まずはサンプルデータを作る。
library(tidyverse) #-----サンプルデータ作成----- x1 = seq(1, 10, by = 1) x2 = seq(12, 20, by = 1) x3 = seq(25, 30, by = 1) x4 = seq(33, 36, by = 1) x5 = seq(46, 50, by = 1) x = c(x1, x2, x3, x4, x5) # A y1 = -(x - 25)^2 + 100 # B y2 = x y2[30] = 100 # C y3 = sin(x/10+10) # D y4 = x + rnorm(length(x), 0, 5) dsamp = data.frame(x = x, y1 = y1, y2 = y2, y3 = y3, y4 = y4)
これらの散布図を描くと次のようになる。
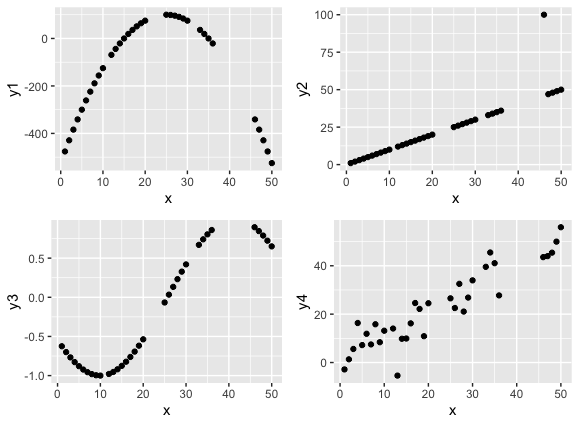
に分割してプロットを配置する方法は下記の記事を参考にさせていただいた。
qiita.com
#-----プロット作成----- p1 = ggplot(dsamp, aes(x = x, y = y1)) + geom_point() p2 = ggplot(dsamp, aes(x = x, y = y2)) + geom_point() p3 = ggplot(dsamp, aes(x = x, y = y3)) + geom_point() p4 = ggplot(dsamp, aes(x = x, y = y4)) + geom_point() #-----分割----- library(gridExtra) grid.arrange(p1, p2, p3, p4)
相関係数を計算してみよう。以前紹介したPearsonの相関係数、Spearmanの相関係数は、いずれも関数corで算出できる。
#-----相関係数の計算----- cor(dsamp$x, dsamp$y1, method = "pearson") cor(dsamp$x, dsamp$y1, method = "spearman") cor(dsamp$x, dsamp$y2, method = "pearson") cor(dsamp$x, dsamp$y2, method = "spearman") cor(dsamp$x, dsamp$y3, method = "pearson") cor(dsamp$x, dsamp$y3, method = "spearman") cor(dsamp$x, dsamp$y4, method = "pearson") cor(dsamp$x, dsamp$y4, method = "spearman")
第1引数・第2引数に、相関をみたい2つの変数を入力する。methodでは、相関係数の種類を指定している。この他にも"Kendall"が指定できるが、これはKendallの順位相関係数(Kendallのタウと呼ばれる)を出力する。
cor関数を使うと、以下のようにシンプルに相関係数のみが出力される。
> cor(dsamp$x, dsamp$y1, method = "pearson") [1] -0.02709525 > cor(dsamp$x, dsamp$y1, method = "spearman") [1] 0.1937161 > > cor(dsamp$x, dsamp$y2, method = "pearson") [1] 0.8931001 > cor(dsamp$x, dsamp$y2, method = "spearman") [1] 0.9969442 > > cor(dsamp$x, dsamp$y3, method = "pearson") [1] 0.9184119 > cor(dsamp$x, dsamp$y3, method = "spearman") [1] 0.842628 > > cor(dsamp$x, dsamp$y4, method = "pearson") [1] 0.9220893 > cor(dsamp$x, dsamp$y4, method = "spearman") [1] 0.9095493
相関係数が0か0じゃないかを検定するcor.testという関数もある。相関係数自体も出力されるので、こちらを使うのもいいかもしれない。
#-----相関係数の検定----- cor.test(dsamp$x, dsamp$y1, alternative = "two.sided", method = "pearson", conf.level = 0.95) cor.test(dsamp$x, dsamp$y1, alternative = "two.sided", method = "spearman", conf.level = 0.95)
最初の2つの引数と、methodの指定はcor関数と同様である。alternativeは両側検定の場合"two.sided"、片側検定で対立仮説が「相関は0より大きい」とする場合には"greater"、「相関は0より小さい」とする場合には"less"を指定する。Pearsonの相関係数のみ信頼区間も計算でき、conf.levelは信頼区間の水準の選択に用いる。
例えば、左上にプロットしたデータのPearsonの信頼区間の検定結果は以下のようになる。
> cor.test(dsamp$x, dsamp$y1, alternative = "two.sided", method = "pearson", conf.level = 0.95)
Pearson's product-moment correlation
data: dsamp$x and dsamp$y1
t = -0.15333, df = 32, p-value = 0.8791
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.3619446 0.3139470
sample estimates:
cor
-0.02709525 p値は0.8791で、相関係数が0であることは否定できていない。
ただ、このような検定はあまりやらないほうがいいかもしれない。左上の図のような二次関数の関係性があるデータは明らかに相関係数のような単調性の指標で測るべきものではないのだが、以下のようにサンプルサイズを際限なく増やせば検定で有意になってしまう。
#-----同様のデータでサンプルサイズを1000倍にする-----
> xx = rep(x, 1000)
> yy = -(xx - 25)^2 + 100
> cor.test(xx, yy, alternative = "two.sided", method = "pearson", conf.level = 0.95)
Pearson's product-moment correlation
data: xx and yy
t = -4.9978, df = 33998, p-value = 5.827e-07
alternative hypothesis: true correlation is not equal to 0
95 percent confidence interval:
-0.03771385 -0.01647052
sample estimates:
cor
-0.02709525相関係数の推定値は-0.027しかないのに、p値は0.0001未満となり、通常使われるどんな有意水準でも相関が0という仮説を否定することになる。下手をすると「直線的な相関があることが統計学的に証明された」という誤解を生みかねないので、少なくとも推定値の大きさ、プロット、そしてどれくらいのサンプルサイズから得られた結果なのかも加味して結果を検討したほうがいいと思う。
まとめ
相関係数をRで計算する方法を紹介した。ここに載せた程度の計算であれば、初期設定で使用できるcor関数やcor.test関数で実施できる。
参考文献
[1] R: The R Stats Package
[2] Schober P. et al. (2018), "Correlation Coefficients: Appropriate Use and Interpretation", Anesth Analg.