傾向スコア法による重み付け解析を行うためのRパッケージいくつか
はじめに
前回の記事では、傾向スコア(Propensity score)による逆確率重み付け法(Inverse probability of treatment weighting; IPTW)をRで行うためにIPWパッケージを用いました。
実は、傾向スコア法による解析を行うためのRのパッケージはたくさんあり、僕自身どれを使えばいいのかいまいちよくわかっていません。
例えば、後述するPSweightパッケージのチュートリアルペーパーでは、重み付けを行えるパッケージだけでも少なくとも9つがあることが述べられています。
そこで今回は、その中で比較的目にすることの多いパッケージ3つを紹介し、サンプルデータを使って基本的な解析の手順を書いていきたいと思います。
なお今回はIPTWの場合に限定しますが、それぞれのパッケージは他の重み付け(例えば、処置群における処置効果(Average treatment effect on the treated; ATT)の推定)にも対応しています。
今回紹介するのは以下のパッケージです。
- WeightIt
- twang
- PSweight

実行例
まずは、今回使用するパッケージを読み込んでおきます。
library(tidyverse) library(broom) library(WeightIt) library(twang) library(PSweight) library(cobalt) library(survey)
- tidyverseは、データの加工やプロットに必須のパッケージです。
- broomはtidyverseに含まれるパッケージですが、別途読み込むことで統計モデルの出力結果をデータフレーム形式(正確にはtibble形式ですが、以降ではデータフレームと呼ぶことにします)に変換するtidy関数が使用できます。
- WeightIt、twang、PSweightは、今回のIPTWの解析に使用する3パッケージです。
- cobaltは傾向スコアによる調整の前と後の共変量のバランスを確認するのに便利なパッケージです。
- surveyは、IPTWにおいて、処置効果の推定量の標準誤差にロバスト分散(Robust variance)を適用するために用います。*1
サンプルデータの説明と未調整の解析
今回は、twangパッケージに付属しているサンプルデータセットの"lindner"を使用します。
data("lindner", package = "twang")データセットの最初の10行は以下のようになっています。

このlindnerデータセットには、経皮的冠動脈インターベンション(PCI)を受けた996名の治療後6ヶ月後の生存状況が記録されています。
また処置変数はアブシキシマブという薬剤の投与有無であり、PCIに加えてこの薬剤を投与することにより、6ヶ月後の生存状況に違いがあるのかを評価したいものとします(参考資料[2]・twangパッケージのチュートリアルペーパーや、参考資料[3]のウェブサイト The Lindner Example を参照)。
今回の解析例では以下の変数を使用します。
- abcix:アブシキシマブ投与の有無(1:あり、0:なし)
- stent:ステント挿入の有無(1:あり、0:なし)
- height:身長(cm)
- female:性別(1:女性、0:男性)
- diabetic:糖尿病の有無(1:あり、0:なし)
- acutemi:最近の急性心筋梗塞の有無(1:あり、0:なし)
- ejecfrac:左室駆出率(%)
- ves1proc:PCIの対象になった血管の数
- sixMonthSurvive:PCI6ヶ月後の生存状況(TRUE:生存、FALSE:死亡)
以下、アブシキシマブ投与ありのグループを処置群、投与なしのグループを対照群とよぶことにします。またPCI6ヶ月後の生存割合をアウトカムとし、処置効果は生存割合の差で評価するものとします。
まずは、共変量の調整なしで処置効果を算出してみます。
今回は生存割合の差*2について、正規分布による近似を考え、線形回帰モデルにより算出しました。
# 共変量調整をしない解析 mod_naive <- glm(sixMonthSurvive ~ abcix, data = lindner, family = gaussian) ## tidy(conf.int = TRUE)により推定値と信頼区間をデータフレーム化 mod_naive %>% tidy(conf.int = TRUE)
abcixの行に、生存割合の差や信頼区間が表示されています。生存割合の差は3.46%(95%CI : 1.30-5.61%)ほどのようです。

しかし、次に見るように両グループの背景因子には偏りが生じており、処置の効果を正しく推定するためには背景因子の調整を行う必要がありそうです。*3
そこで、背景因子の分布について、cobaltパッケージを用いて確認してみます。
cobaltパッケージには、グループ間の背景因子のバランスを数値化して出力するbal.tab関数や、背景因子の偏りの度合いを表す標準化差(Standardized mean difference)を視覚的に表現してくれるlove.plot関数などが搭載されています。
ここではlove.plotを使用してみましょう。
love.plot(abcix ~ stent + height + female + diabetic + acutemi + ejecfrac + ves1proc,
data = lindner, estimand = "ATE",
abs = TRUE, stats = "mean.diffs", thresholds = c(m = 0.1),
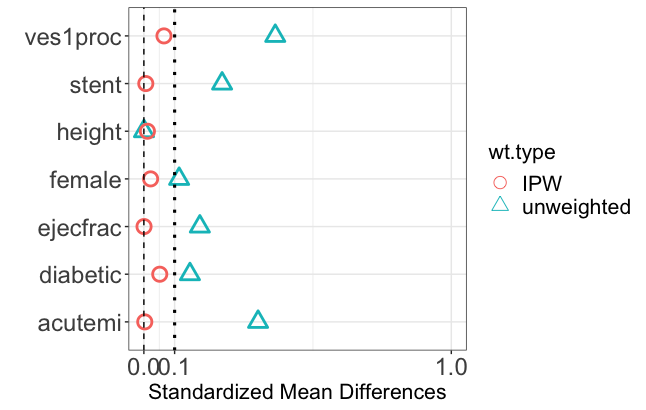
s.d.denom = "pooled", binary = "std")以下の図が出力されます。

縦軸には背景因子、横軸にはそれらの因子の「標準化平均差(Standardized mean difference)」(グループ間の平均値の差を、標準偏差で割って標準化したもの)の絶対値が表示されています*4。この標準化平均差の絶対値は0.1未満になることが好ましいとされていますが、stent、acutemi、ves1procについてグループ間で比較的大きな偏りがあるように見えます。
なお、以降では標準化平均差をSMDと表記します*5。
WeightItパッケージ
では、傾向スコアによるIPTWで解析してみましょう。
まずはWeightItパッケージを使用します。これは傾向スコアの重み付け解析に特化したパッケージで、使われている方も多いかと思います。なお傾向スコアマッチングについてはMatchItというパッケージがあります。
傾向スコアモデルから重みを算出するには、weightit関数を使用します。
まずglm関数などと同様の記法によって傾向スコアのモデル式を定義します。またmethod = "ps"とすることで、ロジスティック回帰によって傾向スコアが推定されます。傾向スコアの推定方法は他にも"generalized boosted modeling"のような機械学習の手法も選択できます。
estimand = "ATE"とすることにより、IPTWに用いる逆確率重みが計算されます。他には"ATT"などの選択肢があります。
# 傾向スコアの推定と重みの算出
psmodel_wtit <- weightit(abcix ~ stent + height + female + diabetic + acutemi + ejecfrac + ves1proc,
data = lindner, method = "ps", estimand = "ATE")上記で得られた結果から、$psで傾向スコアの推定値を、$weightsで重みを取り出すことができます。
# 傾向スコアと重みをもとのデータセットに格納 lindner_iptw_wtit <- lindner %>% mutate(ps = psmodel_wtit$ps, weight = psmodel_wtit$weights)
推定した傾向スコアの両グループでの分布を、cobaltパッケージのbal.plot関数で確認してみましょう。
# propensity scoreの分布 bal.plot(lindner_iptw_wtit, treat = lindner_iptw_wtit$abcix, var.name = "ps")
傾向スコアの分布に重なり合う部分が少ない場合には解析が不適切になる恐れがありますが、今回は大きな偏りはなさそうですので、このまま解析を進めても大丈夫ということにします。

重み付け後の背景因子のバランスも確認してみましょう。
love.plot(abcix ~ stent + height + female + diabetic + acutemi + ejecfrac + ves1proc,
data = lindner_iptw_wtit, weights = lindner_iptw_wtit$weight,
abs = TRUE, stats = "mean.diffs", thresholds = c(m = 0.1),
s.d.denom = "pooled", binary = "std")赤色の点が調整前のSMD絶対値、青色の点が傾向スコアによるIPTWを行った後でのSMD絶対値です。
重み付けを行うことにより、背景因子のバランスが非常に改善されているのが分かります。

続いてIPTWにより生存割合の差を推定するため、surveyパッケージのsvyglm関数によって重み付け解析を行います。解析モデルは調整なしの場合と同様に線形回帰としました。
# 重みやデータの情報を格納
design_wtit <- svydesign(id = ~ 1, weights = ~ weight, data = lindner_iptw_wtit)
# 重み付けの線形回帰
mod_iptw_wtit <- svyglm(sixMonthSurvive ~ abcix,
design = design_wtit, family = gaussian())
# 割合の差の推定値を出力
mod_iptw_wtit %>% tidy(conf.int = TRUE)結果は以下の通りです。生存割合の差は6.61%(95%CI : 0.80 - 12.40%)で、調整しない場合と比べ差が大きくなりました。もともとの背景因子の偏りを考えると、推定にバイアスを生じさせるような交絡がIPTWによって取り除かれた可能性があります*6。

twangパッケージ
続いて、twangパッケージでの解析例です。
twangパッケージの初期バージョンの開発は2004年であり、かなり歴史のあるパッケージです。元々このパッケージは、ロジスティック回帰を用いて最適な傾向スコアモデルを構成する(交互作用や高次の項も含め、どのような説明変数をモデルに入れるかを決定する)作業が非常に負担になっている状況を改善するために、機械学習の手法を用いて自動的に最適な傾向スコアモデルを決定できるようにしたいというのが開発の動機だったようです(参考資料[4]・RAND Corporationのウェブサイト Toolkit for Weighting and Analysis of Nonequivalent Groups (TWANG) | RAND )。
twangパッケージでの傾向スコアの推定は機械学習の手法であるgradient boostingによって行われており、Rでの設定についてもそれらの知識が必要になる箇所が見受けられます。
そのため、この記事内では詳細には触れず、IPTWの解析手順のみ紹介します(力不足で申し訳ありません)。
またチュートリアル(参考資料[2])の例にもあるように、twangによる解析結果はロジスティック回帰により傾向スコアを推定した場合と異なることがあるようです。これは傾向スコア推定方法の違いによるものと考えられます。
古典的なロジスティック回帰とブースティングなどの機械学習手法のどちらを用いるのがよいかは、ここでは一概には言えない難しい問題のように思います。
さて、まずは傾向スコアを推定して、重みを取得します。
# 傾向スコアの推定
psmodel_twng <- ps(abcix ~ stent + height + female + diabetic + acutemi + ejecfrac + ves1proc,
data = lindner, estimand = "ATE", verbose = FALSE)
# 重みをget.weights関数で取得
lindner_iptw_twng <- lindner %>%
mutate(weight = get.weights(psmodel_twng, stop.method = "es.mean"))WeightItと異なり、twangパッケージには傾向スコアや背景因子のバランスを確認するための関数があらかじめ用意されています(ただしcobaltと体裁は異なります)。前述と同様の図を作成するには、以下のコードを実行します。
# propensity scoreの分布 plot(psmodel_twng, plots = 2) # weighting後のバランス plot(psmodel_twng, plots = 3)


重み付け解析については、WeightItの時と同様にsurveyパッケージの関数などを利用する必要があるようです。
# 調整解析
design_twng <- svydesign(id = ~ 1, weights = ~ weight, data = lindner_iptw_twng)
mod_iptw_twng <- svyglm(sixMonthSurvive ~ abcix,
design = design_twng, family = gaussian())
mod_iptw_twng %>% tidy(conf.int = TRUE)結果は以下の通りで、WeightItパッケージで解析した場合と異なり、調整をしない場合とほとんど変わらない推定値になりました。
twangで推定した傾向スコアでは背景因子のバランスが揃えきれていないためか、逆にWeightItでの傾向スコアモデルが妥当でなく(ロジスティックモデルが適切でない、共変量の交互作用などが必要、など)そちらの解析結果が間違っているのかはこれだけでは判断がつきませんが、このようなことが起こりうると考えておいたほうがよさそうです。

PSweightパッケージ
PSweightパッケージは、2020年に登場した比較的新しいパッケージです。
これも傾向スコアによる重み付け解析に特化したパッケージですが、WeightItやtwangと大きく異なるのは、傾向スコア推定と重みの算出、バランス確認、重み付け解析をすべて一つのパッケージの中で完結できる点です。また、最近注目されている重み付け方法であるOverlap weightingにも対応しています*7。
ここではチュートリアルペーパー(参考資料[5] https://cran.r-project.org/web/packages/PSweight/vignettes/vignette.pdf)に沿って、PSweightの使用例を紹介します。
まず最初に、傾向スコアモデルの数式をオブジェクトへ格納します。
数式の表記法はglm関数などと同様のおなじみのものです。
# PSのモデル式 psmodel_pswt <- abcix ~ stent + height + female + diabetic + acutemi + ejecfrac + ves1proc
傾向スコアの分布や重み付け後の背景因子のバランスを確認するには、SumStat関数により傾向スコアの推定を行い、その結果にplot関数を適用します。weight = "IPW"を選択することで逆確率重み付け法(IPTW)の重みが得られますが、他にもoverlap weight(weight = "overlap")やATTを推定するための重み(weight = "treated")などが選択できます。
# 傾向スコアや重みの情報を取得 bal_pswt <- SumStat(ps.formula = psmodel_pswt, data = lindner, weight = "IPW") # 傾向スコアの密度 plot(bal_pswt, type = "density") # 背景因子のバランス plot(bal_pswt, type = "balance")
傾向スコアの分布と背景因子のバランスについて、それぞれ以下のようなプロットが出力されます。
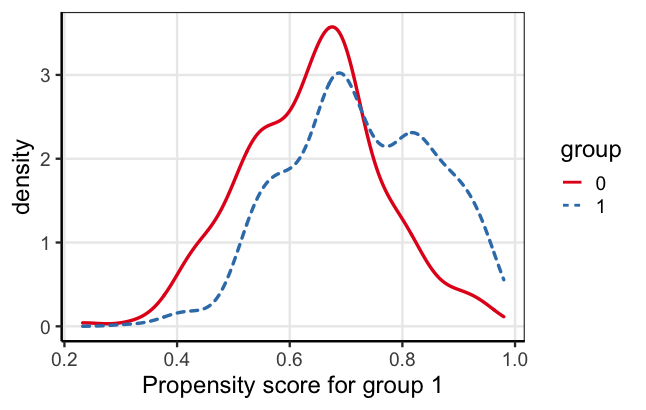
PSweightもロジスティック回帰により傾向スコアを推定しているので、WeightItの場合と同じ結果になっているのが分かるかと思います。
最後にIPTWによる生存割合の差の推定を行います。PSweight関数で推定を行い、summary関数で推定結果を表示します。
なおsummary関数のデフォルトではグループ間の平均の差(二値アウトカムの場合、割合の差)が出力されますが、二値アウトカムの場合にはtype = "RR"と指定するとリスク比が、type = "OR"と指定するとオッズ比が出力されます。
# IPTW
ate_pswt <- PSweight(ps.formula = psmodel_pswt, yname = "sixMonthSurvive", data = lindner,
weight = "IPW")
# 結果の出力(tidy関数には対応していないようです)
summary(ate_pswt, type = "DIF")結果は以下のようになりました。
推定値はWeightItと丸め誤差を除き一致しました。標準誤差の算出にはロバスト分散が用いられていますが、surveyとは算出方法が異なっていると思われ、若干異なった結果になりました。

おまけ:自力で傾向スコアを計算
最後に、多少の手間はかかりますが、自前でロジスティック回帰を用いてIPTWを行う例を紹介します。
バランスの確認や重み付け解析はWeightItパッケージを用いる場合と同様です。
# 傾向スコアの推定
psmodel_man <- glm(abcix ~ stent + height + female + diabetic + acutemi + ejecfrac + ves1proc,
data = lindner, family = binomial)
## 傾向スコア推定値を格納
ps_man <- psmodel_man$fitted.values
# 傾向スコア推定値と逆確率重みをデータフレームへ格納
lindner_iptw_man <- lindner %>%
mutate(ps = ps_man,
weight = case_when(abcix == 1 ~ 1/ps, TRUE ~ 1/(1-ps)))
# IPTW
design_man <- svydesign(id = ~ 1, weights = ~ weight, data = lindner_iptw_man)
mod_iptw_man <- svyglm(sixMonthSurvive ~ abcix,
design = design_man, family = gaussian())
mod_iptw_man %>% tidy(conf.int = TRUE)推定結果はWeightItの場合と全く同じになったので省略します。
まとめ
今回は傾向スコアによる重み付け解析を行うためのRパッケージ3つをとりあげ、IPTWを行う例を簡単に紹介しました。
WeightItは王道的なパッケージのような印象で、解説や使用例はweb上に多く見つかるため、使い勝手がよいように思います。
twangは機械学習の手法を用いて傾向スコアの推定を行う点で、他とは少しテイストが異なるパッケージかもしれません。実用上はロジスティック回帰で傾向スコアを推定することが多いように感じますが、ブースティングをはじめとした機械学習の手法を用いた傾向スコア解析の性能についてはさまざまな研究がなされており、有力な選択肢の一つと考えられます。
PSweightは傾向スコアの推定・バランス評価・重み付け解析までを一つのパッケージ内で数行のコードで完結できるのが魅力的に思います。現時点ではグループ間の平均差やリスク差・リスク比・オッズ比の推定にしか対応していないようですが、今後の機能の拡張に期待したいです。
参考資料
[1] Austin, P. C. (2016). Variance estimation when using inverse probability of treatment weighting (IPTW) with survival analysis. Statistics in medicine, 35(30), 5642-5655.
[2] Ridgeway, G., McCaffrey, D., Morral, A., Cefalu, M., Burgette, L., Pane, J., & Griffin, B. A. (2021). Toolkit for Weighting and Analysis of Nonequivalent Groups: A guide to the twang package. vignette, July, 26.
[3] The Lindner Example
[4] Beth Ann Griffin, Greg Ridgeway, Andrew R. Morral, Lane F. Burgette, Craig Martin, Daniel Almirall, Rajeev Ramchand, Lisa H. Jaycox, Daniel F. McCaffrey. Toolkit for Weighting and Analysis of Nonequivalent Groups (TWANG) Website. Santa Monica, CA: RAND Corporation, 2014. http://www.rand.org/statistics/twang.
[5] Propensity Score Weighting in R: A Vignette.
https://cran.r-project.org/web/packages/PSweight/vignettes/vignette.pdf
*1:IPTWでは、統計モデルから標準的な方法で算出した標準誤差は過小評価(その結果、間違って統計的有意になりやすく、また信頼区間は本来あるべき幅よりも狭くなる)になることが知られています。ただしロバスト分散では逆に過大推定となることがあるため、ブートストラップ法による推定を推奨する意見もあります。(参考資料[1]・Austinの論文)
*2:割合の差は「Risk difference; RD」と呼ばれることが多くありますが、生存する割合をリスクと呼ぶと少し不自然なのでその用語は使いませんでした。
*3:背景因子がアウトカムに影響しうるのかの検討はしていませんが、手法の紹介なので、ここではそういうものとして考えてもらえればと思います。
*4:細かな点はlove.plot関数の設定で変更できます。
*5:Absolute standardized mean differenceの頭文字をとるとASMDですが、あまり使われていないように思いますので、絶対値については「SMDの絶対値」と表記することにします。
*6:あくまで手法の例示のため、具体的な解釈には踏み込まないことにします。
*7:Overlap weightingを実行できるパッケージとして他にはPSWパッケージがありますが、現時点では3グループ以上での解析に対応しているのはPSweightパッケージだけのようです。